This article is available in Czech only. For translation or more information on this topic, please contact author.
Souhrn
Enterokoky patří spolu se zástupci druhu Escherichia coli mezi tzv. indikátory fekálního znečištění, které se používají při hodnocení mikrobiologické kvality koupacích vod. Jejich stanovení se řídí vyhláškou MZ ČR č. 238/2011 Sb. a provádí se kultivačně na selektivních agarových médiích. Je známo, že ne všechny druhy enterokoků jsou fekálního původu a mají tedy přímou souvislost s fekálním znečištěním vody. Pro správné hodnocení kvality koupací vody by bylo vhodné znát i původ těchto bakterií. K trasování původu mikroorganismů (tzv. MST, „microbial source tracking“) se s výhodou používá metod molekulární biologie. Pomocí PCR metody lze amplifikací molekulárních markerů (tj. specifických úseků DNA) odlišit různé druhy enterokoků. Publikací zabývající se touto tématikou je mnoho, nicméně ve většině studií se k druhovému rozlišení používá pouze jeden marker, a to při práci s přírodními vzorky nemusí být vždy dostatečné. Pro spolehlivou druhovou identifikaci u přírodních vzorků by bylo výhodnější použít kombinaci několika markerů. Zároveň by bylo vhodné aplikovat poznatky získané z experimentů s čistými kulturami na přírodní vzorky koupacích vod, a to jak čistých, tak fekálně znečištěných. Pro praxi by byly důležité i postupy umožňující PCR ze směsného přírodního vzorku, tedy bez nutnosti předkultivace na selektivních médiích, aby se maximálně zkrátila doba, za kterou je znám výsledek.
Cílem tohoto příspěvku je přehledně shrnout publikované molekulární markery pro identifikaci jak enterokoků, tak příbuzných mikroorganismů, a zhodnotit jejich možné použití v mikrobiologii vody pro rychlé zařazení zástupců rodu Enterococcus do druhů při analýze přírodních vzorků.
Charakteristické znaky enterokoků
Enterokoky jsou gram pozitivní, kataláza negativní koky uspořádané obvykle do krátkých řetízků. Jsou to chemoorganotrofové, jejichž hlavním koncovým produktem při fermentaci cukrů je kyselina mléčná. Ačkoliv fenotypické a biochemické odlišení enterokoků od ostatních bakteriálních druhů je složité, mezi hlavní znaky tohoto rodu patří přítomnost D antigenu, tolerance k vyšším teplotám (až 45 °C) i schopnost růstu v přítomnosti 6,5% NaCl. Při jejich identifikaci v laboratoři se používají selektivní média, na kterých jsou eliminovány ostatní bakteriální druhy (např. SB médium obsahující azid sodný pro potlačení G- tyček).
Taxonomicky patří mezi Enterococcacae a v současné době zahrnuje tento rod 43 druhů [1]. Typickým zástupcem je E. faecalis. Enterokoky se přirozeně vyskytují v trávicím traktu teplokrevných živočichů včetně člověka i v trávicím traktu některých bezobratlých (korýši, šneci) [2, 3] a byly izolovány i z povrchových sladkých i slaných vod, z půdy či z rostlinné vegetace. Některé druhy (např. E. faecalis, E. faecium) jsou podmíněně patogenní.
Přítomnost enterokoků ve vodě může mít různý původ: buď jsou přirozenou součástí vodního ekosystému a vyskytují se tam volně, nebo mohou být asociovány s rostlinnou vegetací (typicky E. mundtii, E. casseliflavus) [4, 5], nebo se zooplanktonem [6], případně se mohou do vody dostat z půdy [7]. Dalším zdrojem mohou být odpadní vody či exkrementy živočichů, a to jak obratlovců, tak bezobratlých. Za typické intestinální druhy jsou považovány E. faecalis, E. faecium, E. durans, E. hirae [8].
Jak je z výše uvedeného výčtu patrné, ke správné interpretaci zvýšené hladiny enterokoků ve vodním prostředí by bylo vhodné znát jejich původ.
Metody, které se používají k vystopování původu určitých organismů v prostředí, se souhrnně nazývají „microbial source tracking“ (MST). Principem těchto metod je, že určité druhy bakterií jsou asociovány s určitými hostiteli či prostředím. U enterokoků jsou např. druhy E. faecalis, E. faecium považovány za typické intestinální druhy (ačkoliv byly izolovány i z jiných ekosystémů) a naopak E. mundtii, E. casseliflavus jsou většinou spojeny s rostlinami [9, 10]. Jednou z metod MST je PCR amplifikace určitých (definovaných) úseků DNA (tzv. molekulárních markerů), na jejichž základě je možné rozlišit enterokoky do jednotlivých druhů. V tomto směru existuje značné množství literatury týkající se nejen enterokoků, ale i jiných rodů bakterií, a je možné z toho při hledání vhodných molekulárních markerů vycházet.
Molekulární markery pro MST
Obecná charakteristika molekulárních markerů
Vhodný molekulární marker pro druhové rozlišení bakterií by měl splňovat několik kritérií:
- měl by být široce rozšířen v genomech bakterií, tj. ve většině bakterií by měl mít ortologní gen,
- na druhou stranu také musí obsahovat úseky unikátní pro daný druh bez možných paralogních genů,
- jejich velikosti musí být dostatečné pro možná porovnávání, ale zároveň ne příliš dlouhé, aby se daly celé sekvenovat,
- sekvence musí být dostatečně reprezentativní, aby postihla spolehlivě charakteristiku genomu daného druhu.
Tabulka 1. Přehled diskutovaných molekulárních markerů
Table 1. Summary of discussed molecular markers
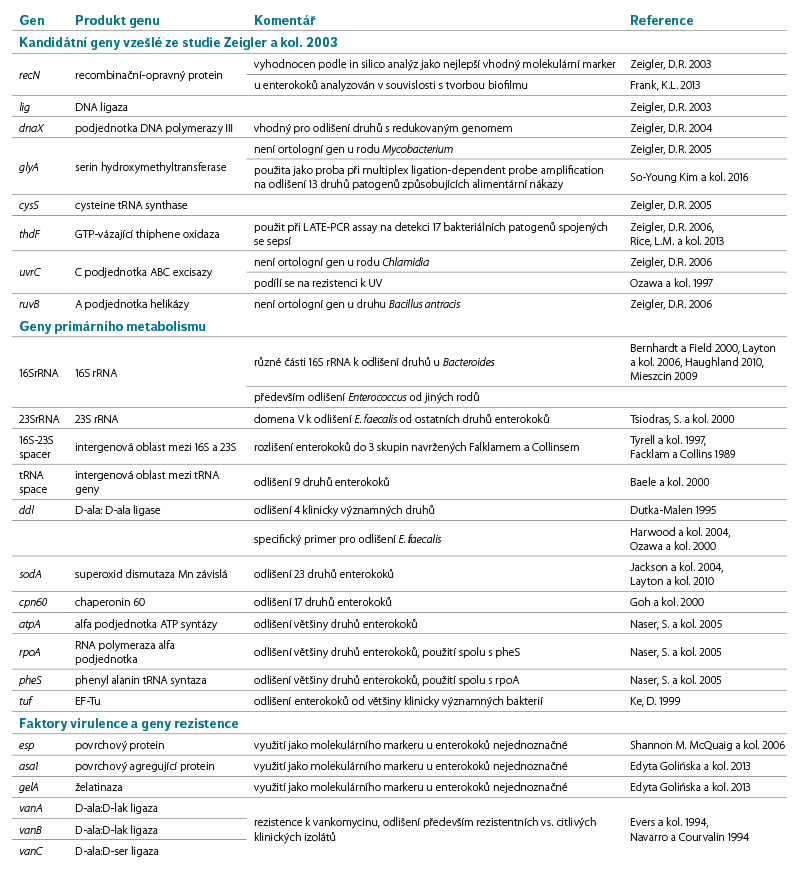
Markery diskutované v tomto příspěvku jsou shrnuty v tabulce 1. Zeigler a kolektiv porovnávali ve své práci 44 bakteriálních genomů, reprezentujících 16 rodů, za účelem hledání takových genových oblastí, které by mohly být použity obecně na rozlišení bakteriálních druhů. Z celkových analýz nakonec vyplynulo 8 různých genů (genových oblastí), které splňovaly výše uvedená kritéria [11]. Daná studie nezahrnovala genom enterokoků, avšak pracovala mimo jiné s genomy 4 druhů streptokoků, mezi něž se enterokoky původně řadily. Z navržených 8 kandidátních genů byly 4 (recN, glyA, thdF, uvrC) zkoumány i u enterokoků, a to buď v souvislosti s tvorbou biofilmu (recN), nebo resistence k UV (uvrC) [12, 13]. Geny glyA a thdF byly použity spolu s jinými geny pro odlišení různých bakteriálních patogenů způsobujících alimentární infekce [14, 15]. Amplifikace recN byla použita i pro odlišení několika druhů streptokoků [16]. Tento gen se tedy nabízí jako jeden z několika vhodných kandidátů též k detekci enterokoků.
Skupiny genů vhodných pro MST
Geny kódující komponenty transkripčního a translačního aparátu
Geny, jejichž produkty se podílí na expresi genetické informace, tedy na transkripci a translaci, splňují podmínku přítomnosti ortologů (tj. ve všech mikroorganismech, kde jsou přítomné, plní stejnou funkci) a zároveň též obsahují úseky specifické pro daný druh. Proto také 16S a 23S rDNA byly mezi prvními kandidáty na molekulární markery pro MST. Použitím různých oblastí 16S rDNA jako biologického markeru se zabývalo mnoho skupin, a to nejen u enterokoků [17], ale v souvislosti s MST též u bakterií rodu Bacteroides [18, 19]. Bacteroides tvoří významnou část střevní mikroflóry teplokrevných živočichů včetně člověka, a proto o nich také bylo uvažováno jako o možných alternativních indikátorech fekálního znečištění [20, 21]. Na rozdíl od E. coli a intestinálních enterokoků jsou ale obtížně kultivovatelné. Jejich zástupci jsou druhově specifičtí pro určité hostitele [22], čehož by bylo možné s výhodou využít právě pro MST. Sekvenace 16S rRNA patří stále mezi nejpoužívanější metody pro identifikaci druhů enterokoků, přestože má svoje limity při rozlišení blízce příbuzných druhů, zejména ze skupiny E. faecium [23].
Tsiodras s kolegy analyzoval sekvence genu 23S rDNA kódujícího RNA velké podjednotky bakteriálního ribozomu. Použili doménu V 23S rDNA pro odlišení E. faecalis od ostatních druhů enterokoků [24].
Později si badatelé všimli, že zatímco geny pro 16S a 23S rRNA jsou fylogeneticky velmi konzervované, intergenové oblasti vykazují větší variabilitu a byly by tedy pro odlišení druhů vhodnější. Na základě srovnání intergenové oblasti mezi 23S a 16S rRNA Tyrell a kolektiv mohli rozlišit enterokoky do tří fylogenetických skupin (I, II, III, navržených dříve Facklamem a Collinsem) [25, 26]. V rámci skupiny II bylo navíc i možné odlišit intestinální druh E. faecium od typicky přírodního druhu E. casseliflavus a druhu E. mundtii. Výhodou tohoto markeru je též fakt, že pro amplifikaci se pro všechny druhy používá jeden pár primerů a jednotlivé druhy se liší velikostí amplifikovaného úseku (úseků).
Obdobně Baele a kolektiv použili intergenovou oblast mezi geny pro tRNA k odlišení devíti druhů enterokoků [27]. Při těchto experimentech použili primery navržené v krajních konzervovaných oblastech tRNA genů, pro amplifikaci oblasti mezi těmito geny. To by mohlo umožnit využití této strategie u mnoha bakteriálních druhů, čemuž nasvědčuje fakt, že obdobná genová oblast byla použita pro druhové rozlišení u streptokoků [28], Acinetobacter spp. [29], Listeria spp. [30] a stafylokoků [31, 32].
Dalším slibným markerem je gen rpoA kódující α podjednotku RNA polymerázy. Bylo ukázáno, že při použití rpoA spolu s dalším markerem – pheS (gen pro fenylalanyl-tRNA syntázu) je možné rozlišit 30 druhů enterokoků [33]. Gen rpoA byl též mezi kandidátními geny navrženými Zeiglerem a kol. nicméně s menším skóre než výše zmíněných vybraných osm kandidátních genů. Zdá se však, že pro enterokoky by mohl být na odlišení druhů spolehlivě použit.
Podobně lze gen tuf kódující elongační faktor Tu využít pro odlišení enterokoků od ostatních klinicky významných bakterií [34]. Je třeba ale podotknout, že navržené primery se vázaly též na sekvence ortologních genů u několika druhů Listeria spp. a zástupců rodu Abiotrophia.
Geny kódující další proteiny primárního metabolismu
Poroznost je věnována i genům kódujícím proteiny primárního metabolismu, především genům ddl, sodA, atpA a cpn60.
Gen ddl kóduje D-ala:D-ala ligázu nezbytnou při syntéze peptidoglykanu. Na rozdíl od amplifikace intergenových oblastí, při použití genu ddl, byla použita pro různé druhy různá dvojice primerů. Výsledkem PCR byl potom buď specifický namnožený úsek DNA odpovídající genu daného druhu, nebo žádný produkt. Nevýhodou této metody je, že v případě většího množství druhů znamená identifikace tímto způsobem velké množství jednotlivých PCR reakcí. Použitím ddl jako markeru bylo možné rozlišit čtyři druhy enterokoků (E. faecium, E. faecalis, E. gallinarum, E. casseliflavus) [35]. Harwood a kolektiv později identifikovali ddl úsek, který byl specifický pro odlišení E. faecalis od ostatních druhů. Nicméně odlišení druhého typického intestinálního druhu E. faecium bylo s tímto markerem dost složité a často ani nekorelovalo s výsledky biochemických testů [36].
Na obdobném principu, tedy navržení dvojice primerů specifických pro daný druh v rámci jednoho genu navrhli Jackson a kolektiv 23 párů primerů pro amplifikaci genu sodA kódující superoxiddismutázu závislou na Mn [37]. Tyto primery byly pak použity hromadně v celkem 7 PCR reakcích (multiplex PCR). Layton a kolektiv navíc optimalizovali pozici některých primerů, a tím zvýšili jejich druhovou specifitu [38].
Produktem genu atpA je alfa podjednotka ATP syntázy, tedy enzymu zodpovědného za syntézu ATP. Bylo zjištěno, že tímto markerem je možné rozlišit většinu druhů enterokoků. Pomocí atpA spolu s rpoA a pheS bylo možné ještě zvýšit spolehlivost druhového určení a navrhnout kombinaci této trojice markerů spolu s 16S rDNA jako vhodné geny pro typizaci enterokoků [33, 39].
Goh a kolektiv využili degenerovaných primerů k odlišení 17 druhů enterokoků na základě amplifikace genu pro chaperonin (cpn 60) [40].
Geny pro faktory virulence a antibiotické resistence
Je na místě se domnívat, že intestinální druhy enterokoků se mohou od těch volně v přírodě žijících druhů lišit produkcí faktorů virulence včetně produkce adhesinů. Ačkoliv se původně myslelo, že např. povrchový protein Esp s funkcí adhesinu je specifický pro izoláty E. faecalis pocházejících z intestinálního traktu člověka [41], později byl tento protein nalezen i u izolátů ze zvířat či jiných přírodních vzorků [42, 43].
Přítomnost genů pro jiné faktory virulence jako např. pro povrchový agregující protein (asa 1) či gen pro želatinázu (gel E) se lišila i u klinických izolátů jednoho druhu [44]. Z tohoto důvodu by mohlo být použití těchto genů jako markerů nejednoznačné.
Typicky přírodní druhy enterokoků (E. casseliflavus a E. gallinarum) jsou, na rozdíl od druhů izolovaných z klinických vzorků, obecně citlivé k antibiotikům. Geny rezistence se často používají jako markery pro odlišení bakteriálních druhů i rezistentních versus citlivých izolátů především v humánní medicíně. K největším současným problémům antibiotické rezistence u enterokoků patří rezistence k vankomycinu, který byl dlouho podáván při infekcích multirezistentními kmeny především u hospodářských zvířat. Vankomycin inhibibuje syntézu peptidoglykanu, na jehož syntéze se mimo jiných podílí též výše diskutovaná D-ala:D-ala ligáza (gen ddl). Geny, jejichž produkty jsou zodpovědné za rezistenci k vankomycinu, vanA, vanB, vanC-1, a vanC-2 jsou příbuzné k ddl [35, 45, 46]. Proti vankomycin rezistentním enterokokům se v současné době používá antibiotikum linezolid inhibující proteosyntézu. Na resistenci k linesolidu se podílí produkty genů cfr kódující ribozom modifikující enzymy a optrA, jehož produktem je ABC transporter [47]. Avšak použití genů rezistence pro rozlišení humánních a zvířecích druhů má svoje limity – geny rezistence se mohou v populaci rychle šířit horizontálním přenosem nebo spolu s transpozičními elementy a jejich přenos úzce souvisí se selekčním tlakem v prostředí. Navíc izoláty enterokoků humánního původu mohou vykazovat značné rozdíly v citlivosti k antibiotikům i v rámci jednoho druhu a jednoho prostředí. Proto použití antibiotických genů jako markerů má pravděpodobně jen omezené uplatnění.
Závěr
Příspěvek shrnuje testované a publikované molekulární markery, které byly nebo mohou být použity pro druhové rozlišení enterokoků, což by mělo pomoci při charakterizaci jejich původu v koupacích vodách. Většina studií se zabývá vždy jedním vhodným genem, ale to může být při analýze přírodních navíc znečištěných vzorků nedostačující. Pro spolehlivé zařazení enterokoků do druhů by bylo tedy lepší použít kombinaci několika genů včetně jednoho rodově specifického obsahujícího část 16S rRNA. Naser navrhl použít kombinaci genů rpoA, pheS atpA a 16S rDNA pro typizaci enterokoků. Svoje výsledky však ověřoval na čistých kulturách. Bylo by tedy zajímavé přenést tyto poznatky do praxe a ověřit je i na přírodních vzorcích koupacích vod. Kromě toho by z uvedeného výčtu mohl být pro typizaci použit ještě některý z genů navržených Zeiglerem a kolektivem jako např. gen recN případně glyA nebo thdF. Pro tyto geny by ale bylo nutné udělat ještě podrobnější in silico sekvenační analýzy. Zdá se, že kromě kódujících oblastí, jsou zajímavé i intergenové oblasti, jako např. oblasti mezi geny pro tRNA. Jejich výhodou je možnost použití universálních primerů v konzervovaných oblastech i možnost srovnání s jinými rody, u nichž byl již tento přístup použit.
Výčet zde prezentovaných kandidátů není zdaleka úplný a v literatuře existuje ještě řada dalších možných genových oblastí, které by bylo možné použít jako např. další geny kódující proteiny primárního metabolismu, nebo naopak repetitivní nic-nekodující sekvence. Některé nebyly uvedeny z praktického důvodu, neboť se nehodily pro jednoznačné odlišení původu znečištění nebo k nim není v současné době dostatek dat (nebylo na ně odkázáno ve více studiích). Je zde však velký potenciál dalších možných kandidátů.
Poděkování
Tento příspěvek vznikl v rámci projektu TA ČR TJ04000132 Využití metod molekulární biologie k identifikaci zdrojů znečištění v koupacích vodách.
